O código a seguir é um exemplo de execução de KNN no R. Além do código para executar o algoritmo em si, você verá que há uma parte de análise descritiva que eu inseri. É sempre interessante saber o que há no seu conjunto de dados. No caso, utilizaremos um dataset famoso chamado iris que não requer nenhum download. Peço encarecidamente que você visite o post Algoritmo de Classificação: KNN (K Nearest Neighbors), caso conheça pouco do algoritmo KNN.
Bom, vamos começar carregando a base e analisando o que há dentro dela.
# Mostra as primeiras linhas
head(iris)
# Mostra as ultimas linhas
tail(iris)
# Resumo (minimo, maximo, mediana, quartis)
summary(iris)
# Resumo 2 (quantidade, missing, quartis, etc)
library("Hmisc");
describe(iris);
# Estrutura das variaveis (tipo)
str(iris)
# Histograma de cada variavel por especie
library("ggplot2")
ggplot(iris,aes(x=Sepal.Length))+geom_histogram()+facet_grid(~Species)+theme_bw()
ggplot(iris,aes(x=Sepal.Width))+geom_histogram()+facet_grid(~Species)+theme_bw()
ggplot(iris,aes(x=Petal.Length))+geom_histogram()+facet_grid(~Species)+theme_bw()
ggplot(iris,aes(x=Petal.Width))+geom_histogram()+facet_grid(~Species)+theme_bw()
# Graf de Dispersao por especie
qplot(Sepal.Length, Sepal.Width, data=iris, color=Species)
qplot(Petal.Length, Petal.Width, data=iris, color=Species)
Este começo é bastante autoexplicativo, então não vejo muita necessidade de abordar estes trechos iniciais. O que estou fazendo aí é apenas entendendo o que temos em mãos, não diz respeito especificamente ao knn. Como podemos ver, temos uma base com as informações dos comprimentos e larguras das pétalas e caules de cada espécie de flor. O que queremos é, com esse histórico, conseguir prever a espécie de uma flor com base nas informações de pétala e caule. Tendo esta base em mãos, se alguém te trouxer uma flor com a pétala tendo comprimento 1.5 e largura 0.5 e de caule com comprimento 5.0 e largura 3.6, você consegue dizer qual a espécie dessa flor? Por enquanto, imagino que não. Mas acompanhe as linhas a seguir que você será capaz disso.
Como expliquei no post Algoritmo de Classificação: KNN (K Nearest Neighbors), o KNN faz uso da distância entre os pontos. Então precisamos ajustar as grandezas das medidas:
# Scale data # Scale: (x - media(x)) / desvpad(x) iris_normal = iris iris_normal[, -5] = scale(iris[, -5])
Como também já foi explicado no post mencionado, podemos usar o histórico para fazer predições. Porém, não adianta a gente tentar rodar um modelo com essa base inteira, a gente precisa usar parte dela para criar o modelo e a outra parte para testá-lo. Logo, a gente deve separar a base em treino e teste. Vamos reservar 60% da base para treinar o modelo, i.e. para criar nossas classes com base nas informações e os 40% restantes usaremos para testar a acurácia do modelo.
set.seed(000000) # Separa em treino e teste ## Gera indices da base treino e teste train_index = sample(1:nrow(iris_normal), 0.6*nrow(iris_normal), replace = FALSE) ## Gera base treino e teste treino = data.frame() treino = iris_normal[train_index,] teste = data.frame() teste = iris_normal[-train_index,]
Agora, o que fazemos é rodar o algoritmo. O que o R irá fazer é usar a base treino para definir as classes (no caso as espécies) e usar essa informação para predizer a que classe pertence cada flor da base teste. Depois, verificamos o acerto do modelo:
# Carrega biblioteca para rodar KNN
library("class")
# Roda com k=2
Knn_K2= knn(treino[,-5], teste[,-5],
treino$Species, k=2, prob=TRUE)
# Roda com k=3
Knn_K3= knn(treino[,-5], teste[,-5],
treino$Species, k=3, prob=TRUE)
Testei o modelo para k igual a 2 e 3. Era possível testar para outros casos, mas por enquanto vou ficar somente nesses dois. Vejamos qual teve maior acerto:
# Analisa a matriz de confusao para cada um table(teste$Species, Knn_K2) table(teste$Species, Knn_K3) # Analisa acuracia sum(Knn_K2==teste$Species)/length(teste$Species)*100 sum(Knn_K3==teste$Species)/length(teste$Species)*100

A matriz de confusão mostra qual a espécie que o modelo previu e qual era a correta. É interessante pois você pode observar quanto de erro tipo I e tipo II seu modelo gera. No caso da acurácia, verificamos o acerto como um todo. Pela acurácia, o melhor modelo seria o utilizado para k = 3.
Claro que agora você está pensando “po, mas testar cada k ali é muito chato, por que não automatizar”? E foi isso que um usuário do Kaggle (Xavier Vivancos García) fez:
# Método para comparar diferentes k's
# cria uma lista para receber as predicoes
Knn_Testes = list()
# cria variavel para receber acuracia
acuracia = numeric()
# cria loop para testar de k=1 ate k=20
for(k in 1:20){
Knn_Testes[[k]] = knn(treino[,-5], teste[,-5], treino$Species, k, prob=TRUE)
acuracia[k] = sum(Knn_Testes[[k]]==teste$Species)/length(teste$Species)*100
}
# Comparacao grafica das acuracias
plot(acuracia, type="b", col="blue", cex=1, pch=1,
xlab="k", ylab="Acuracia",
main="Acuracia para cada k")
# Linha vertical vermelha marcando o maximo
abline(v=which(acuracia==max(acuracia)), col="red", lwd=1.5)
# Linha horizontal cinza marcando o maximo
abline(h=max(acuracia), col="grey", lty=2)
# Linha horizontal cinza para marcar o minimo
abline(h=min(acuracia), col="grey", lty=2)
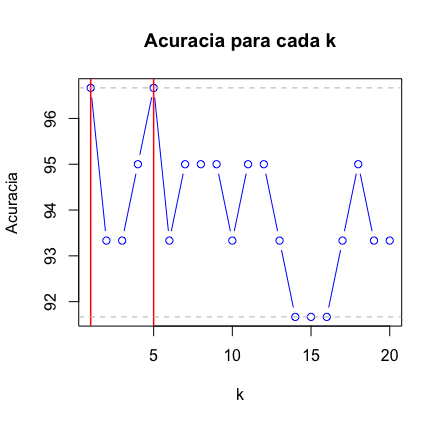
Se você simplesmente digitar acuracia no R você vai obter a acurácia para os 20 casos. Fique a vontade para rodar para quantos k’s achar necessário. Como pudemos verificar, para k=1 e k=5, temos uma boa acurácia. Se utilizarmos k=5, podemos então fazer a previsão para aquela flor que queríamos lá em cima? Aquela com a pétala tendo comprimento 1.5 e largura 0.5 e de caule com comprimento 5.0 e largura 3.6. Bom, para isso, o que precisamos fazer é substituir essa flor pela base teste na execução do KNN:
# flor que queremos definir a especie flor_exemplo = c(5, 3.6, 1.5, 0.5) # copia o cabecalho da base teste base_exemplo = teste[0:1, -5] # une cabecalho com a flor base_exemplo = rbind(base_exemplo, flor_exemplo) # exclui primeira linha base_exemplo = base_exemplo[2,] Knn_K5_Predicao = knn(treino[,-5], base_exemplo, treino$Species, k=5, prob=TRUE) Knn_K5_Predicao
Pronto, aí está a espécie da flor! Bom, deve ser possível criar o dataframe a partir do teste[] de um jeito mais inteligente do que a maneira demonstrada acima (confesso que me causou certo desconforto). De qualquer forma, espero que o código seja proveitoso para você.
E se você gostou do post, não vá embora sem deixar uma curtida ou um comentário. Eu sei que não parece relevante, mas faz diferença para mim e custa pouco para você, vai… Se encontrou algum erro ou tem alguma sugestão, dúvida, elogio ou crítica, pode escrever nos comentários ou me enviar uma mensagem diretamente em Sobre o Estatsite. E visite também a conta do Twitter @EstatSite, costumo postar algumas coisas bem rapidinho por lá, geralmente são posts e códigos mais curtos ou compartilhamento de algo legal ou alguma reflexão.
Ótimo resumo da aplicação! Obrigado!