Análogo ao que foi feito no post Machine Learning do Zero no Python, teremos agora no blog um tutorial bem tranquilo, do ZERO, para quem quer aprender Machine Learning / Ciência de Dados, utilizando R. Não fique com medo por ser um post mais extenso, você pode fazer um pouco cada dia durante um certo período de tempo, caso sinta que está pesado demais. Abaixo, você vai aprender a tratar os dados, fazer filtros, localizar valores nulos, criar gráficos e até conhecer alguns modelos. Então bora aprender!
1. JÁ POSSUI O R INSTALADO?
Antes de qualquer coisa, instale o RStudio e esteja minimamente familiarizado com seu funcionamento. Para isso, vá até o tutorial Primeiros passos em R (Studio). Não há segredo nenhum, lá explico até como instalar o software. Creio que em menos de 1h você terá terminado de ler o post. Mas é claro, não se espante se levar mais tempo.
2. CARREGANDO E ENTENDENDO A BASE
Vamos começar com você baixando a base que eu vou utilizar neste tutorial. Ela é uma pequena modificação do dataset iris, que já vem no próprio R. Fiz algumas alterações para que a gente pudesse realizar algumas tarefas que seriam impossíveis com a base original. Já explico as informações que existem nela. Para baixar a versão que criei, Iris_Alterada.csv, descompacte o zip e salve aonde achar conveniente.
Começamos importando a base a ser utilizada:
# carrega Iris_Alterada em uma base que voce nomeou como dados dados = read.csv(file = 'Iris_Alterada.csv', header=TRUE, sep=',', dec = '.')
Primeira lição aqui, para importar os dados em csv, utilize read.csv. Caso tenha cabeçalho, use o argumento header = TRUE, caso contrário, header = FALSE. O argumento sep serve para indicar qual o separador. Normalmente é ‘;’, porém, no meu caso era somente a vírgula. Já o argumento dec serve para dizer qual o símbolo para o decimal. Via de regra, bases de países como EUA utilizam ponto, no Brasil utilizamos vírgula. O símbolo do jogo da velha (a hashtag) serve para fazer comentários. SEMPRE comentem seus códigos. De verdade, você não vai saber o que cada trecho significa depois de algumas semanas.
ATENÇÃO: O R é o que chamamos de “case sensitive”. Isto é, é diferente você escrever read.csv() e Read.csv(). Na verdade, o segundo sequer funcionaria. Esteja atento para a utilização de letras maiúsculas ou minúsculas.
Agora, você provavelmente quer saber o que há nessa base. Aqui, há duas opções e a sua escolha depende principalmente do tamanho da base. Você pode verificar só algumas linhas para ver se tudo foi carregado sem erros, e também se você entende o conteúdo dela:
# verifica as primeiras linhas head(dados) # Verifica as ultimas linhas tail(dados)
Isso é o que você vai enxergar como saída:
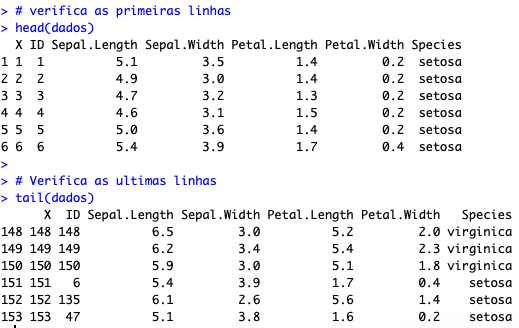
Como você pode ver, ali estão as primeiras e últimas linhas da sua base. A base é formada por informações de flores. Temos 7 colunas apresentadas ali:
- X: Coluna que não serve para nada, é apenas a contagem de linhas da base;
- ID: Um código de identificação única para identificar qual a flor. Seria como um RG para as flores da amostra;
- Sepal.Length: Comprimento da sépala da flor;
- Sepal.Width: Largura da sépala da flor;
- Petal.Length: Comprimento da pétala da flor;
- Petal.Width: Largura da pétala da flor;
- Species: Nome da espécie.
Ou seja, levantaram uma amostra de 153 flores, contendo suas características e o nome da espécie. Por qual motivo? Bom, talvez alguém quisesse entender as características visíveis aos olhos que determinam qual a espécie da flor, entender como identificar a flor com base nisso. Imagine que ao invés das características da flor, fossem o endereço e a profissão de um indivíduo; e ao invés da espécie, tivéssemos a marcação de que o indivíduo pagou ou não a dívida com o banco. Isso serviria para que um cientista de dados estudasse quais as profissões e bairros que tendem a pagar a dívida. Esses são alguns dos exemplos mais comuns de estudos feitos por cientistas de dados.
Bom, agora, vamos imaginar que você esteja acostumado a ver a base por completo e não somente algumas linhas. Nesse caso, você pode usar a função View(), que abrirá uma nova aba no seu RStudio. O problema aqui é que nem sempre tem utilidade, já que você não vai conseguir ficar olhando linha a linha, e nem é essa a tarefa do cientista de dados. E também podemos enfrentar problemas se for uma base muito grande. Sendo assim, minha recomendação é que você se acostume com head() e tail().
Inclusive, falando de funções, caso você tenha alguma dúvida de como alguma função funciona, além (é claro!) do Google, você pode utilizar o help(). Veja no exemplo com head() e o print de todo meu RStudio para ajudar no entendimento:
help(head)
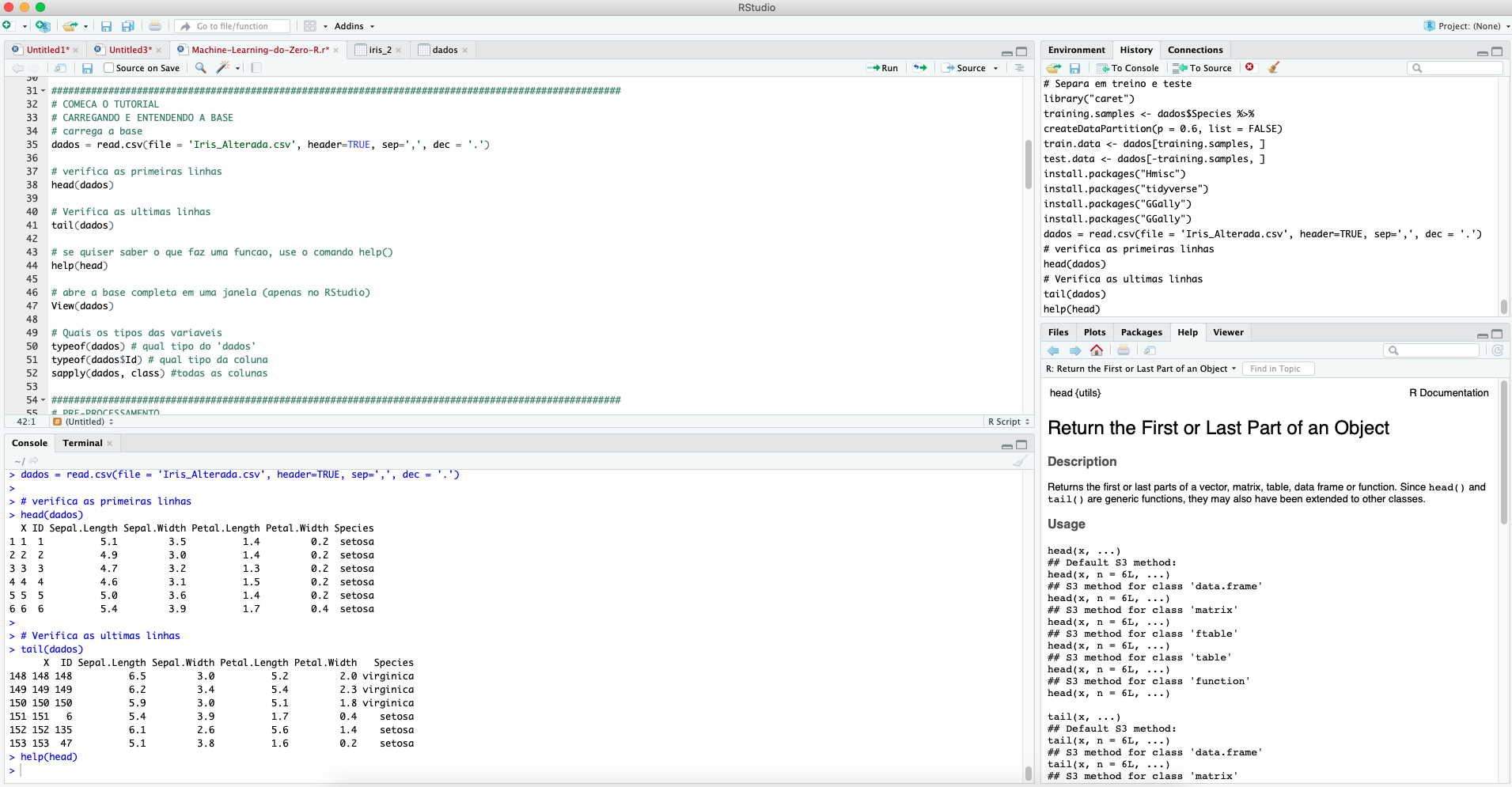
Veja que no canto inferior direito abriu uma explicação sobre a função. Se você descer mais a barra de rolagem dessa janela contendo as informações da função head(), você verá até alguns exemplos de uso da função descrita.
Bom, agora que você carregou os dados, seria interessante entender o que há lá dentro, quais os tipos de variáveis que você está lidando. Veja 3 exemplos desse tipo de exploração:
# qual tipo do 'dados' typeof(dados) # qual tipo da coluna ID typeof(dados$ID) #todas as colunas sapply(dados, class)
Foram geradas 3 saídas. A primeira, nos mostra que o objeto dados é uma lista. A segunda, mostra que a coluna ID está no formato integer (= inteiro). Por fim, temos a função sapply() que, combinada com o argumento class, traz o tipo de cada variável da base dados.
Aqui vai uma breve explicação sobre o que é cada um dos três formatos da nossa base e também alguns outros comuns que você provavelmente terá que lidar:
- Integer: Valores numéricos inteiros. Exemplo: Número da residência, número de frutas num cesto, número de sapatos comprados no último ano, etc;
- Numeric: Valores numéricos decimais. Exemplo: Altura, saldo bancário, etc.;
- Factor: Variáveis categóricas. Exemplo: Sexo (masculino ou feminino), classe social (A, B, C, D ou E), grau de dificuldade (alto, médio, baixo). É importante entender que o R tratará de forma diferente uma variável numérica que for do tipo factor de outra do tipo numeric. Por exemplo, se você estiver identificando a variável sexo como sendo 1 para mulher e 0 para homem, não faz sentido você tirar a média dela. Logo, se ela estiver devidamente tratada como fator, a função mean() retornará um erro. Esteja atento ao formato da variável;
- Logical: Variável do tipo lógico é normalmente utilizada para expressar uma comparação. Exemplo: a variável z = x > y é uma variável do tipo logical. Ela retorna TRUE ou FALSE.
- String/Character: Variáveis textos, expressadas entre aspas simples ou dupla. Exemplo: Nome do país.
Outra coisa que você observou, creio eu, é que é possível acessar a coluna utilizando o símbolo de ‘$’. Por exemplo, acessamos a coluna ID da base dados quando escrevemos dados$ID. Veja alguns exemplos de como você pode acessar a informação da base:
# acessa a terceira linha da coluna Species dados$Species[[3]] # acessa linha 3 e coluna 4 da base dados[3,4] # acessa a coluna Species dados['Species'] # acessa a linha 3 da coluna Species dados['Species'][[3]] # acessa primeira coluna da base dados[1] # acessa primeira linha da base dados[1,]
Caso tivéssemos um vetor, a ideia seria semelhante. Veja um exemplo simples:
# cria um vetor x = c(1,2,3,3,3,1,1) # acessa quarto elemento do vetor x[4] # exclui o elemento 2 do vetor x[-2]
Agora que você já sabe como funciona o acesso às informações de uma base e um vetor, vamos trabalhar um pouco essa informação. Aliás, já tenha em mente que nenhuma informação vem redonda para o cientista de dados. Vamos colocar a mão na massa pra valer agora!
3. PRÉ-PROCESSAMENTO DA BASE
Agora, vamos ao que é normalmente chamado de pré-processamento da base. É a parte em que tratamos as informações, arrumamos campos que podem conter erros, criamos novos campos caso a gente precise.
Primeiro, o nome da coluna ID veio maiúscula nessa base. Porém, em todas as outras ela está com o ‘i’ maiúsculo e o ‘d’ minúsculo. Para manter o padrão, e já sabendo como acessar informações, você faz uma pequena alteração no nome da coluna:
# Altera nome da coluna names(dados)[names(dados) == "ID"] = "Id"
Se quiser verificar se a alteração funcionou, você pode obter os nomes das colunas com a função names() ou então com colnames(). Não se esqueça que o nome da base deve ser o input desses comandos, ou seja, você deve digitar names(dados) ou colnames(dados).

Já que tudo correu bem, seguimos tratando as informações. Como vimos, a coluna 1 não serve para nada. Podemos excluí-la utilizando o sinal de negativo de forma análoga à seção anterior:
dados[-1]
Funcionou? Bom, apareceu a base inteira sem a primeira coluna. Então funcionou, certo?
Utilize o comando head(dados) e veja você mesmo:
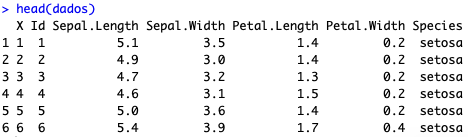
A primeira coluna continua ali!!!
Pois é, a questão é que ela só mostrou o resultado do comando, mas não alocou a informação em nenhum tipo de objeto. Para ‘guardar’ a base sem a primeira coluna, precisamos atribuir o resultado do comando dados[-1] a um objeto. Poderíamos criar uma base nova, uma versão 2 da que temos, como por exemplo dados_v2. Eu gosto de criar versões para acompanhar as mudanças, ainda que isso consuma um pouco de memória. Porém, aqui vamos atribuir a mudança à própria base dados:
dados = dados[-1]
Dessa vez, a alteração deu certo. O próximo passo então é entender se os dados estão corretos. Um problema clássico é termos duplicidades. Sabemos que na nossa base, cada linha representa um indivíduo. Sendo assim, não podemos ter duas linhas iguais. Para verificar a existência dessa duplicidade e tratar da maneira correta, podemos fazer duas ações:
# Localiza Duplicidade da linha inteira duplicated(dados) #aponta linhas duplicadas
sum(duplicated(dados)) # traz a contagem de linhas duplicadas
A primeira ação feita foi verificar quais linhas estão duplicadas. A função duplicated() retorna FALSE para linhas únicas e TRUE caso a linha seja uma cópia de outra. Ou seja, se a linha 5 for igual a linha 1, a linha 5 retornará TRUE. Note que obtivemos 2 valores TRUE, para a última e antepenúltima linha. A segunda ação, foi contar quantas linhas estão duplicadas. Algo muito mais interessante caso a base seja gigante.
Agora que sabemos que essas linhas estão erradas, podemos simplesmente excluí-las:
# remove linhas duplicadas dados = dados[!duplicated(dados), ]
Agora, se você prestou atenção às explicações anteriores, notou outro problema. Nós temos chave única de identificação, o Id. Sendo assim, se tivermos dois campos Id iguais, a nossa informação veio com um erro. Para verificar duplicidade de um campo específico, faremos algo semelhante ao que fizemos acima:
duplicated(dados$Id) # aponta duplicados sum(duplicated(dados$Id)) # traz a contagem de duplicados
Como pudemos ver, temos um caso de duplicidade no campo Id. Para resolver isso, é preciso entender qual o motivo do erro e qual está correto. Aí a solução não é genérica e cada caso é um caso. No nosso, conversamos com o dono da base e ele nos disse que, dentre esses dois Id, o segundo é o que está errado. Então, excluímos da mesma forma que foi feita anteriormente, porém especificando que estamos tirando duplicidade somente para Id:
dados = dados[!duplicated(dados$Id), ] # remove duplicidade do campo Id
Feito esse pequeno tratamento, podemos explorar algumas possibilidades. Por exemplo, imagine que possa existir um Id duplicado, desde que para espécies diferentes. Nesse caso, você pode querer criar uma coluna combinando Id e nome da espécie. Ou então, por algum motivo, você quer combinar valores. Talvez você precise do produto da largura e comprimento da pétala. Para esses dois casos, é simples construir novas colunas:
# Cria nova coluna concatenando campos dados$coluna_nova1 = paste(dados$Id,dados$Species) # cria nova coluna somando campos dados$coluna_nova2 = dados$Petal.Length+dados$Petal.Width # verifica alteracoes head(dados)
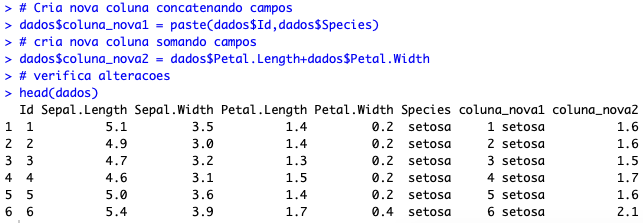
Parece que deu tudo certo. Porém, a gente não precisa realmente disso, foi apenas para mostrar como se cria novas colunas a partir de outras (algo MUITO comum de se fazer).
Vamos então excluir as duas colunas criadas e também o Id, já que agora esse campo não vai nos interessar mais (no dia à dia ele é importantíssimo!).
# Remove coluna criada agora e Id(nao precisaremos mais) dados = dados[c(-1,-7, -8)] # verifica alteracao head(dados)
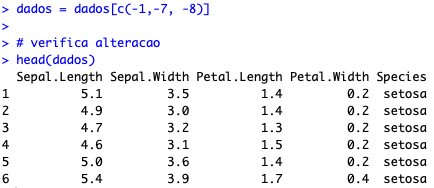
4. ANÁLISE DESCRITIVA (E INSTALAÇÃO DE PACOTES)
Já que limpamos nossa base, vamos entender o que está nela de uma forma mais quantitativa. Vamos obter as estatísticas dela. Mais uma vez, vou colocar duas opções aqui. Primeiro, a função ‘de fábrica’ do R, summary():
summary(dados)
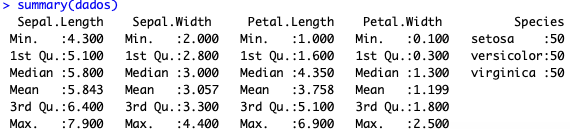
Como vocês podem ver, temos o mínimo, o primeiro quartil, a mediana, a média, o terceiro quartil e o valor máximo para cada variável numérica. Para a variável factor, a categórica, temos a contagem dos dados.
Além dessa função, existe a describe() do pacote Hmisc. E agora você tem DUAS lições: como instalar um pacote e como usar a função describe:
# instala pacote
install.packages("Hmisc");
library("Hmisc");
# aplica describe nos dados
describe(dados);
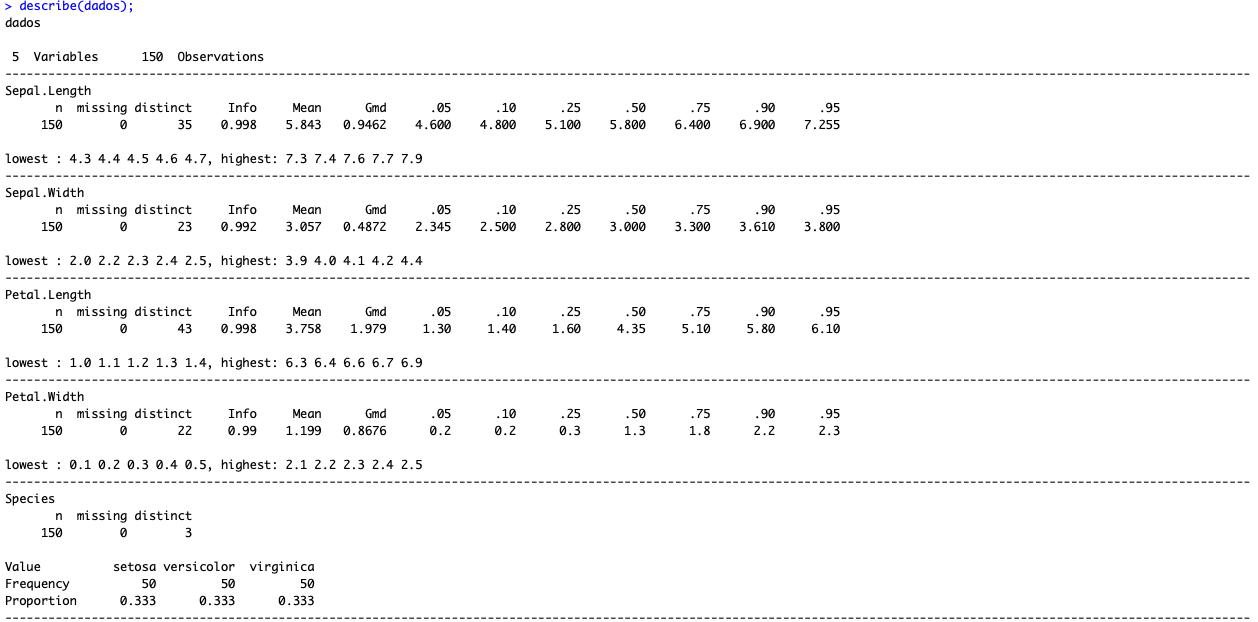
Note que a função describe traz diversos percentis, variáveis missing, contagem e a proporção para o caso das variáveis categóricas. Em outras palavras, é muito mais completa e, portanto, recomendada.
Examinando alguns dos campos apresentados:
- N: Para todas as variáveis, temos 150 elementos/valores preenchidos;
- Missing: Nenhuma variável veio com campo missing (não preenchido);
- Distinct: Para Sepal.Length, temos 35 valores distintos. Já para Sepal.Width, somente 23. Ou seja, para Sepal.Length, os valores se repetem menos;
- .95: Somente 5% das flores possuem Sepal.Length superior a 7.255;
- Mean: A média da largura das pétalas das flores é de 1.199;
Para mais informações sobre percentis e estatística descritiva, leiam Estatística Descritiva e Percentil – Conceito e Código SAS. Em tempo, esses dois posts são de tempos em que o site era menos organizado, então peço desculpa caso você tenha qualquer confusão. Fique a vontade para me escrever.
5. ANÁLISE EXPLORATÓRIA (~ ANÁLISE GRÁFICA)
Agora que já sabemos algumas informações, que tal visualizá-las?
Começamos com um simples histograma de uma das variáveis:
hist(dados$Sepal.Length)
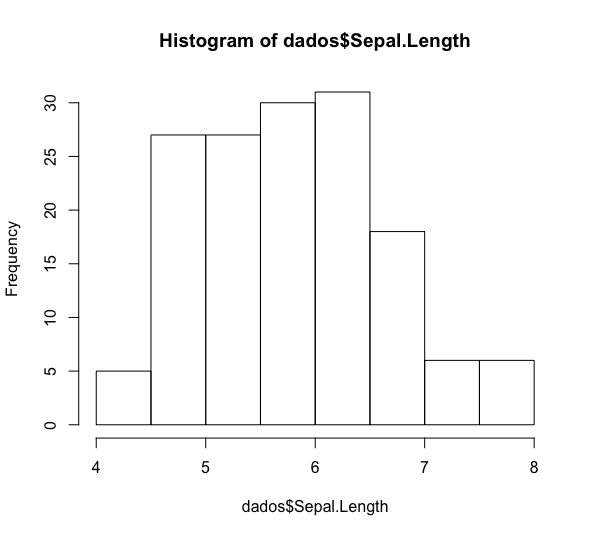
Claro que esse gráfico não está apresentável. Difícil imaginar alguém chegando em uma reunião com ele. Que tal se a gente pintar de uma cor, colocar um título e mudar os nomes dos eixos?
# histograma colorido e com outros detalhes histograma = hist(dados$Sepal.Length, breaks = 10, col="darkblue", xlim=c(4,8),main="Histograma Sepal Length", xlab="Sepal Length", ylab= "Frequencia");
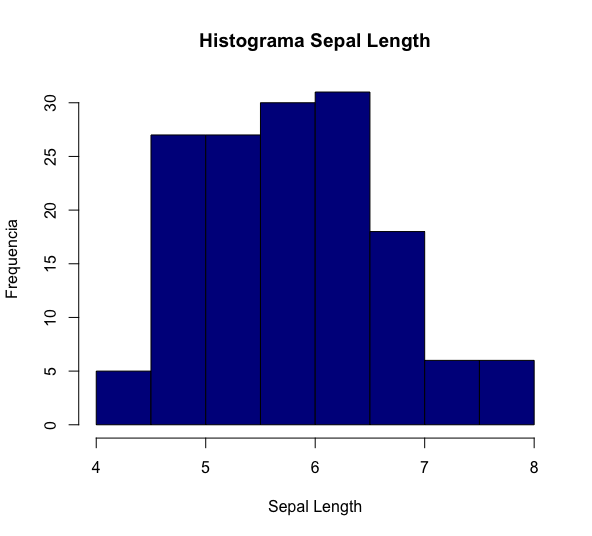
Note que mantivemos o uso da mesma função, hist(), mas com a inclusão de alguns argumentos:
- breaks: número de barras/colunas;
- col: cor do preenchimento;
- xlim: limites do eixo x;
- main: título do gráfico;
- xlab: título do eixo x;
- ylab: título do eixo y.
Um hábito importante, principalmente para os próximos itens, é de utilizar o comando dev.off() uma vez que o gráfico já tiver sido utilizado. Isso serve para encerrar o instrumento gráfico, “limpar” os gráficos do R. Para um gráfico sozinho não costumamos ter nenhum problema, mas para gráficos em painéis, mais bem trabalhados, podemos ter complicações.
É possível também apresentar vários gráficos ao mesmo tempo, em painéis:
dev.off() #encerrar graf anterior # grafico com varios paineis, aqui em uma linha e duas colunas # define como vai ser painel (1 linha, 2 colunas) par(mfrow=c(1,2)) # plota graficos hist(dados$Sepal.Length) hist(dados$Sepal.Width)
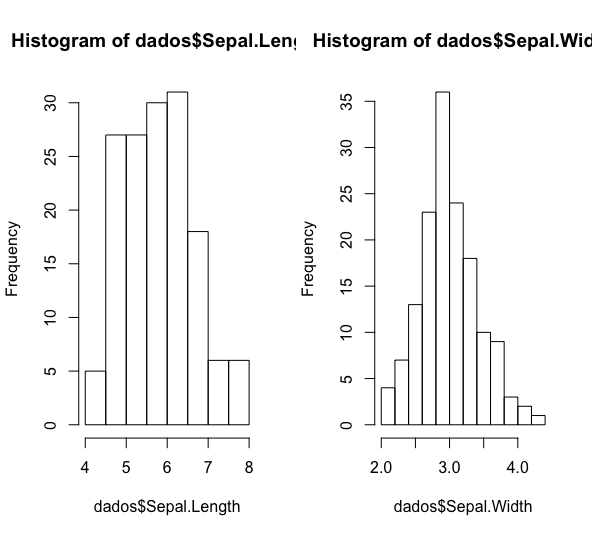
dev.off() #encerrar graf anterior # define como vai ser painel (2 linhas, 1 coluna) par(mfrow=c(2,1)) #plota graficos hist(dados$Sepal.Length) hist(dados$Sepal.Width)
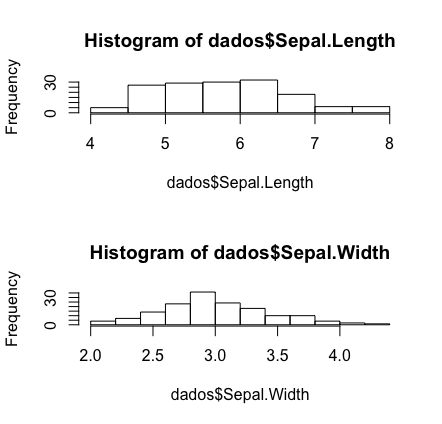
Como já mostrei a forma de melhorar esses gráficos, alterando o título, cor e os nomes dos eixos, fica como dever de casa melhorar esses dois painéis que criamos.
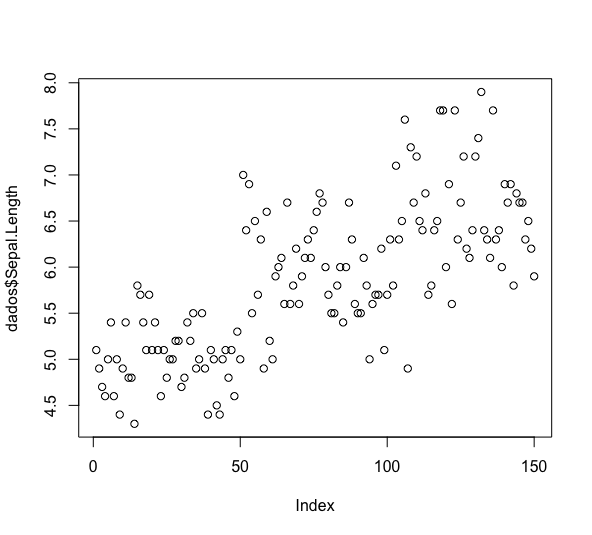
Outro gráfico comumente utilizado é o de dispersão. Novamente, começamos com uma única variável:
dev.off() #encerrar graf anterior plot(dados$Sepal.Length)
Podemos, mais uma vez, fazer algumas mudanças no gráfico. Além do título principal e dos eixos, vamos fazer um gráfico com cada eixo representando uma variável e também vamos traçar uma reta que melhor representa o comportamento dos pontos:
# grafico de dispersao com duas variaveis e linha da regressao plot(dados$Sepal.Length, dados$Sepal.Width, xlab="Sepal Length", ylab = "Sepal Width", main = "Grafico Length x Width") abline(lm(Sepal.Width~Sepal.Length, data=dados), col="red")
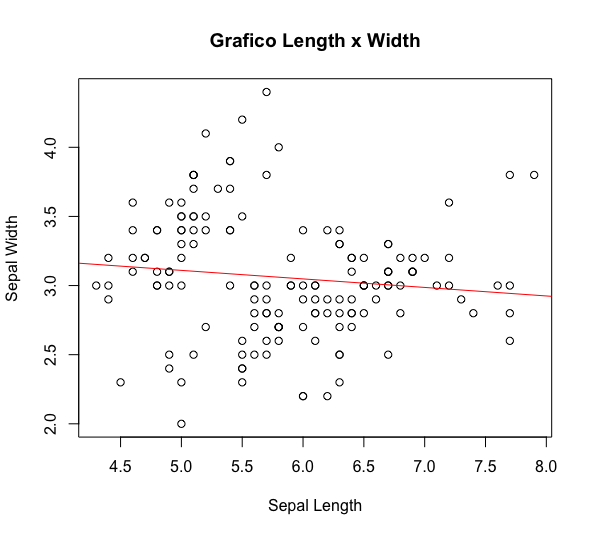
Note que abline está traçando a reta da regressão linear das variáveis em questão. Isso serve para se ter uma ideia do comportamento dos dados, embora não sirva para justificar muita coisa.
Feitas essas representações, o provável último passo do seu projeto será criar modelos preditivos. Em uma empresa, seu modelo poderá servir para prever se o cliente irá pagar ou não uma dívida, ou então saber qual o valor da dívida, ou quem sabe indicar o filme que o cliente mais irá gostar de assistir. Enfim, há várias possibilidades quando tratamos de modelos preditivos. Abaixo, você verá 3 possibilidades. No blog, há muitas outras, como por exemplo Árvore de Decisão no R e Regressão Diff-In-Diff com Efeitos Fixos no R.
6. MODELAGEM ESTATÍSTICA: KNN
Normalmente, inicia-se o estudo de modelos pela Regressão Linear. No entanto, como para Regressão Linear e Logística não usaremos os dados de iris, então deixa-as para o final. Iniciaremos com uma técnica de clusterização chamada KNN. Caso você tenha zero conhecimento em KNN, uma técnica de clusterização bastante utilizada, por favor acesse Algoritmo de Classificação: KNN (K Nearest Neighbors) e volte após concluir a leitura.
O que queremos aqui é, com a informação dessa amostra de flores, conseguir prever a espécie de uma flor com base nas informações de pétala e caule. Ou seja, sabendo o que sabemos a respeito de algumas flores, tendo esta base em mãos, se alguém te trouxer uma flor com a pétala tendo comprimento 1.5 e largura 0.5 e de caule com comprimento 5.0 e largura 3.6, você consegue dizer qual a espécie dessa flor? Por enquanto a resposta é não, mas ela pode se tornar simples se aplicarmos o KNN.
Como foi explicado no post Algoritmo de Classificação: KNN (K Nearest Neighbors), o KNN faz uso da distância entre os pontos para então separar os indivíduos em grupos semelhantes. Creio que agora fique claro, entendendo a técnica, que o que faremos aqui será separar as flores em grupos semelhantes, sendo que cada grupo representará uma espécie. Primeiro, precisamos ajustar as grandezas das medidas:
# Scale data # Scale: (x - media(x)) / desvpad(x) iris_normal = iris iris_normal[, -5] = scale(iris[, -5])
Como também já foi explicado no post mencionado, podemos usar o histórico para fazer predições. Porém, não adianta a gente tentar rodar um modelo com essa base inteira, a gente precisa usar parte dela para criar o modelo e a outra parte para testá-lo. Logo, a gente deve separar a base em treino e teste. Vamos reservar 60% da base para treinar o modelo, i.e. para criar nossas classes com base nas informações e os 40% restantes usaremos para testar a acurácia do modelo.
set.seed(000000) # Separa em treino e teste ## Gera indices da base treino e teste train_index = sample(1:nrow(iris_normal), 0.6*nrow(iris_normal), replace = FALSE) ## Gera base treino e teste treino = data.frame() treino = iris_normal[train_index,] teste = data.frame() teste = iris_normal[-train_index,]
Agora, o que fazemos é rodar o algoritmo. O que o R irá fazer é usar a base treino para definir as classes (no caso as espécies) e usar essa informação para predizer a que classe pertence cada flor da base teste. Depois, verificamos o acerto do modelo:
# Carrega biblioteca para rodar KNN
library("class")
# Roda com k=2
Knn_K2= knn(treino[,-5], teste[,-5],
treino$Species, k=2, prob=TRUE)
# Roda com k=3
Knn_K3= knn(treino[,-5], teste[,-5],
treino$Species, k=3, prob=TRUE)
Testei o modelo para k igual a 2 e 3. Era possível testar para outros casos, mas por enquanto vou ficar somente nesses dois. Vejamos qual teve maior acerto:
# Analisa a matriz de confusao para cada um table(teste$Species, Knn_K2) table(teste$Species, Knn_K3) # Analisa acuracia sum(Knn_K2==teste$Species)/length(teste$Species)*100 sum(Knn_K3==teste$Species)/length(teste$Species)*100

A matriz de confusão mostra qual a espécie que o modelo previu e qual era a correta. É interessante pois você pode observar quanto de erro tipo I e tipo II seu modelo gera. No caso da acurácia, verificamos o acerto como um todo. Pela acurácia, o melhor modelo seria o utilizado para k = 3.
Claro que agora você está pensando “po, mas testar cada k ali é muito chato, por que não automatizar”? E foi isso que um usuário do Kaggle (Xavier Vivancos García) fez:
# Método para comparar diferentes k's
# cria uma lista para receber as predicoes
Knn_Testes = list()
# cria variavel para receber acuracia
acuracia = numeric()
# cria loop para testar de k=1 ate k=20
for(k in 1:20){
Knn_Testes[[k]] = knn(treino[,-5], teste[,-5], treino$Species, k, prob=TRUE)
acuracia[k] = sum(Knn_Testes[[k]]==teste$Species)/length(teste$Species)*100
}
# Comparacao grafica das acuracias
plot(acuracia, type="b", col="blue", cex=1, pch=1,
xlab="k", ylab="Acuracia",
main="Acuracia para cada k")
# Linha vertical vermelha marcando o maximo
abline(v=which(acuracia==max(acuracia)), col="red", lwd=1.5)
# Linha horizontal cinza marcando o maximo
abline(h=max(acuracia), col="grey", lty=2)
# Linha horizontal cinza para marcar o minimo
abline(h=min(acuracia), col="grey", lty=2)
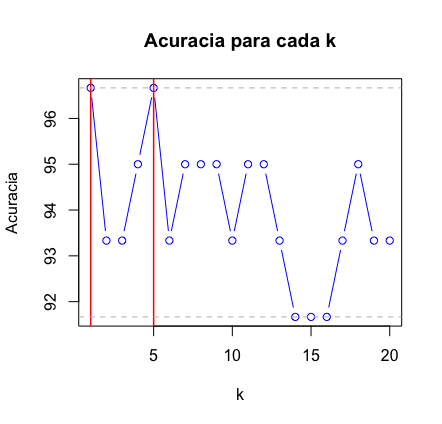
Se você simplesmente digitar acuracia no R você vai obter a acurácia para os 20 casos. Fique a vontade para rodar para quantos k’s achar necessário. Como pudemos verificar, para k=1 e k=5, temos uma boa acurácia. Se utilizarmos k=5, podemos então fazer a previsão para aquela flor que queríamos lá em cima? Aquela com a pétala tendo comprimento 1.5 e largura 0.5 e de caule com comprimento 5.0 e largura 3.6. Bom, para isso, o que precisamos fazer é substituir essa flor pela base teste na execução do KNN:
# flor que queremos definir a especie flor_exemplo = c(5, 3.6, 1.5, 0.5) # copia o cabecalho da base teste base_exemplo = teste[0:1, -5] # une cabecalho com a flor base_exemplo = rbind(base_exemplo, flor_exemplo) # exclui primeira linha base_exemplo = base_exemplo[2,] Knn_K5_Predicao = knn(treino[,-5], base_exemplo, treino$Species, k=5, prob=TRUE) Knn_K5_Predicao
Pronto, aí está a espécie da flor! Bom, deve ser possível criar o dataframe a partir do teste[] de um jeito mais inteligente do que a maneira demonstrada acima (confesso que me causou certo desconforto). De qualquer forma, espero que o código seja proveitoso para você.
7. MODELAGEM ESTATÍSTICA: REGRESSÃO LINEAR
Agora, vamos ao que seria normalmente o primeiro modelo ensinado, a Regressão Linear. Assim como a Regressão Logística, não faremos uso da base iris, pois não faria muito sentido.
A Regressão Linear é uma técnica que nos ajuda a encontrar uma relação entre duas variáveis. Vamos supor que a gente imagine que a relação de salário e anos de estudos seja linear. Em nossa cabeça, quanto mais anos de estudos, maior o salário. Mas como se dá essa relação?
Bom, é aí que entra a Regressão Linear Simples. Utilizando essa técnica, podemos encontrar uma função que descreva como anos de estudos impactam o salário. A forma mais usual de se encontrar essa função é minimizando a soma dos erros ao quadrado. Ficou confuso, né? É tranquilo.
Criamos um gráfico de dispersão onde simulamos uma amostra de dados a respeito de salário e anos de estudos. Ou seja, seria como se tivéssemos coletado dados de alguns trabalhadores, suas informações de salário e anos de estudos, e traçamos um gráfico com esses dados.
A partir da amostra de trabalhadores que usamos para construir o gráfico, queremos um modelo que seja capaz de prever qual o salário de um trabalhador, dado o número de anos de estudos dele. Repare abaixo que as variáveis parecem ter uma relação linear forte. Quanto mais anos de estudos, maior o salário. Parece que podemos traçar uma reta para representar bem essa relação, certo?
Vejamos algumas possibilidades:
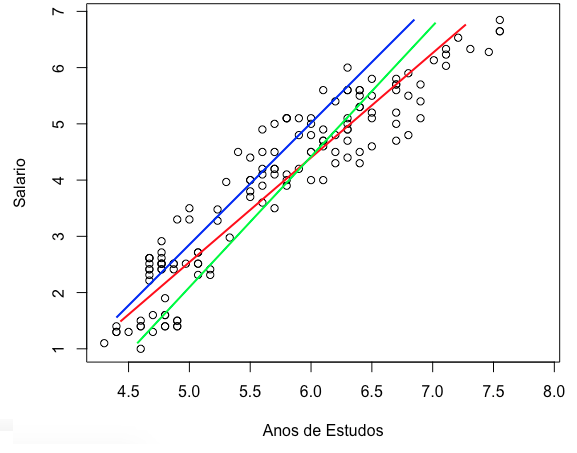
As três retas passadas parecem boas representações da relação entre as nossas duas variáveis. Qual seria a ideal?
Precisamos definir qual a melhor reta, qual a função (=equação da reta) que vai representar a relação entre salário e anos de estudos. O método mais comum é o dos mínimos quadrados. Para entender o método, você precisa primeiro saber o que são os resíduos do modelo. Resíduos nada mais são do que a diferença entre o valor projetado no modelo (na função / equação que estamos tentando descobrir) e o valor real. Ou seja, se a reta traçada prediz que para 6 anos de estudos, o trabalhador deve ter 4 mil reais de salário, mas o ponto observado na amostra foi de 3,5 mil reais, então o resíduo é de 500. No gráfico abaixo, as retas laranjas representam parte dos resíduos:
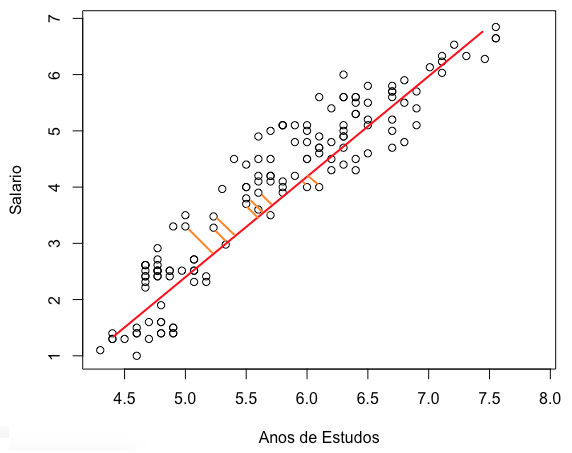
Os resíduos seriam as linhas laranjas para todos os pontos. Ficou claro?
O método dos mínimos quadrados nos diz que a reta a ser escolhida é a que possuir a menor soma dos resíduos ao quadrado. Ou seja, a soma das distâncias entre os pontos reais e a reta, deve ser a menor possível. Essa é a melhor reta, segundo o método dos mínimos quadrados. Se quiser reforçar esse conceito, leia este pequeno artigo.
Caso ainda esteja com dificuldade em entender uma regressão linear, tente estudar mais este material aqui, da Khan Academy.
Veja de cara a diferença dessa técnica para o KNN que vimos acima. Antes, nossa variável resposta era uma classe (a espécie da flor), agora, é uma variável numérica contínua. Um exemplo em que a Regressão Linear pode ser calculada é na tentativa de prever o preço de uma casa com base nas informações dela (número de quartos, se tem ou não jardim, etc.), ou então prever qual deve ser o limite do cartão de crédito de um cliente com base nas informações de renda e idade.
Agora que você entende com clareza, vamos ao exemplo pratico no R.
A base a ser utilizada é simples, é a cars que já vem no R e possui as informações de velocidade de um carro e a distância necessária para que ele pare. Duas variáveis numéricas, contínuas:
# verifica algumas linhas da base head(cars)
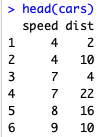
Uma das premissas para que a gente possa utilizar uma regressão linear é que as variáveis possuem uma relação linear entre si. Vamos ver se é o nosso caso fazendo duas coisas:
# plota o grafico das duas variaveis plot(cars$dist,cars$speed)
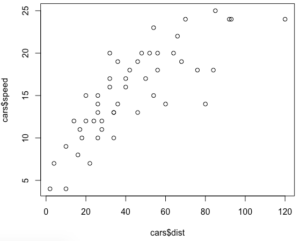
É possível notar que há uma certa linearidade na relação entre as duas.
Podemos ainda calcular a correlação entre ambas, que deve ser bem alta:
cor(cars$speed, cars$dist)
![]()
Sim, é bem alta.
Vamos então construir nossa regressão linear. Para isso, utilizamos a função lm(), onde passamos a variável resposta, seguida do símbolo ‘~’ e as variáveis explicativas. Aqui, queremos tentar prever qual a distância necessária para o carro parar, dada uma determinada velocidade:
# constroi o modelo linearMod = lm(dist ~ speed, data=cars) # exibe os resultados print(linearMod)
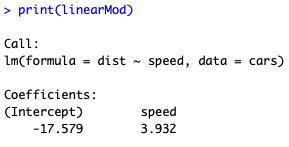
O resultado acima é uma equação, que pode ser escrita da seguinte forma:
distancia = -17.579 + 3.932 * velocidade
Ou seja, se você quiser saber qual a distância necessária para um carro que está a 100mph parar, basta você substituir o valor 100 pela variável velocidade. A resposta seria que você precisaria de 375,621 milhas.
Além de obter a equação, você pode extrair outras informações da sua regressão:
summary(linearMod)
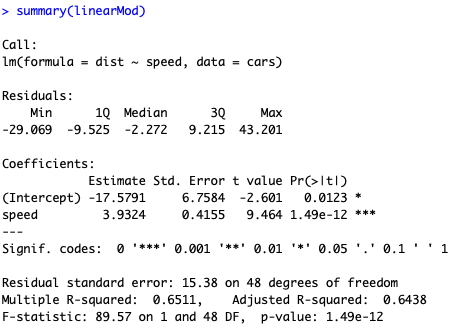
Agora você possui o p-valor para cada variável, o que permite você saber se aquele coeficiente é significativo ou não. E, provavelmente a segunda informação mais observada, você também tem o R-Quadrado. O R-Quadrado indica quanto da variância da variável explicativa (distância) é explicada pelo modelo.
ATENÇÃO: Nesse exemplo, a base não foi dividida em treino e teste para que a gente ganhasse tempo no tutorial. Porém, fica de exercício para casa você rodar a regressão e aplicar o modelo da base treino na base teste.
8. MODELAGEM ESTATÍSTICA: REGRESSÃO LOGÍSTICA
Antes de iniciarmos a construção do modelo, se você possui pouco conhecimento em regressão logística, comece lendo o post Regressão Logística: Conceitos Essenciais e Modelo. Ao terminar, retorne para este post e prossiga com seus estudos.
Vamos construir nossa regressão logística utilizando como exemplo os dados de spam. Inicialmente, vamos carregar o pacote kernlab e os dados de spam:
install.packages("kernlab", dependencies=TRUE)
library("kernlab")
data(spam)
Como você pode não saber exatamente o que há dentro de spam, é importante verificar algumas coisas, como as primeiras linhas da tabela, o total de linhas e colunas, uma estatística descritiva resumida:
head(spam) dim(spam) summary(spam)
Como você pode ver, temos 58 colunas e 4.601 linhas. A base tem diversas características de e-mails (e.g.: quantidade de letras maiúsculas, quantidade de ponto e vírgula, etc.) e uma coluna marcando se o e-mail é um spam ou não. Ou seja, temos um histórico de e-mails, as informações deles e se o e-mail é um spam ou não. Com isso, podemos tentar construir um modelo que seja capaz de prever se um e-mail é um spam ou não, com base em algumas características.
Em primeiro lugar, precisamos separar a base treino e teste. Para facilitar o trabalho, vamos carregar o pacote caret que possui várias funções de modelagem. Em seguida, utilizamos a função createDataPartition para separar a base de forma aleatória na proporção 75-25. Com base na separação feita, criamos a base treino e o que não estiver nela será a base teste:
library(caret); indice.treino = createDataPartition(y=spam$type, p=0.75, list=FALSE) treino = spam[indice.treino, ] teste = spam[-indice.treino, ]
Você pode, e deve, fazer algumas análises na sua base. Por exemplo, temos algum missing nela?
sapply(treino, function(x) sum(is.na(x)))
Note que, para nossa sorte, não há nenhum NA na base inteira. Se houvesse, poderíamos trata-los de diversas formas: excluindo as linhas, preenchendo-as com a média da coluna a qual pertence, preenchendo-a através do método KNN, etc.
Poderíamos, também, fazer uma análise gráfica. Porém, você pode ver diversos tutoriais de gráficos no R e aplicá-los aqui (veja Gráficos em R, Personalizando seu gráfico do ggplot2 – Exports and Imports, William Playfair, Mais gráficos no R: qqplot(), Histograma no R).
Vamos então ao modelo:
modelo = glm (type_new ~ our+over+remove, data = treino, family = binomial) summary(modelo)
Veja que as todas as variáveis são estatisticamente significantes. Você poderia ainda utilizar o stepwise na seleção de variáveis, bastaria rodar step(modelo). No stepwise, as variáveis vão sendo testadas e mantidas de acordo com sua significância.
Se você quiser ver os coeficientes da regressão:
summary(modelo)$coefficients
Lembre-se, os coeficientes de uma regressão logística não são como os de uma regressão linear. Enquanto na regressão linear tínhamos algo como y = beta0 + beta1*x, na regressão logística temos log(p/1-p) = beta0 + beta1*x. Não é necessário, mas você poderia, por causa disso, calcular agora o odds ratio (veja também Regressão Logística: Primeiros Passos):
odd.ratio = exp(coef(modelo))
Seria um outro jeito de ler a equação.
Agora, usamos a função predict para aplicar o modelo em nossa base teste e já aproveitamos para criar uma nova base teste com as projeções feitas pelo modelo:
pred.Teste = predict(modelo,teste, type = "response") Teste_v2 = cbind(teste,pred.Teste)
Para saber se o modelo está bom, vamos usar a curva ROC e o KS:
library(ROCR) pred.val = prediction(pred.Teste ,Teste_v2$type) # calculo da auc (area under the curve) auc = performance(pred.val,"auc") # Plota curva ROC performance = performance(pred.val, "tpr", "fpr") plot(performance, col = "blue", lwd = 5) #Calculo Estatística KS ks <- max(attr(performance, "y.values")[[1]] - (attr(performance, "x.values")[[1]])) ks
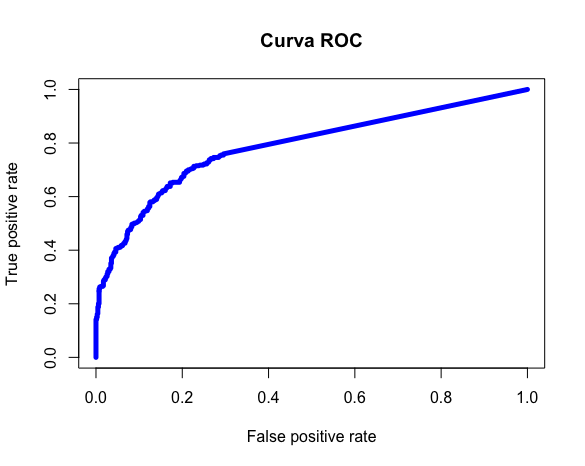
Um KS de 0.48 e uma curva ROC com AUC de 0.78 tornam nosso modelo ótimo.
Poderíamos usar uma matriz de confusão também, porém precisaríamos definir o corte usado na projeção para dizer se o e-mail é spam ou não. Aqui, vamos trabalhar com 0.5:
#confusion matrix table(teste$type, pred.Teste > 0.5)

Dos 697 nonspam, o modelo previu corretamente 655. Dos 352 que eram spam, o modelo acertou corretamente 189.
EXTRA: CONSTRUÇÃO DE IRIS_ALTERADA
Para quem quiser saber como eu criei a base alterada, este é o código (há funções interessantes sendo utilizadas, recomendo o estudo do trecho abaixo):
# cria um dataset com base no iris iris_2 = iris # cria uma coluna de indice (um ID) iris_2$ID = seq.int(nrow(iris_2)) # reordena a coluna iris_2 = iris_2[,c(6,1,2,3,4,5)] # verifica as primeiras linhas (validacao) head(iris_2) # cria linhas extras que serao incluidas linha_extra_1 = c(6, 5.4, 3.9, 1.7, 0.4, 'setosa') linha_extra_2 = c(135, 6.1, 2.6, 5.6, 1.4, 'setosa') linha_extra_3 = c(47, 5.1, 3.8, 1.6, 0.2, 'setosa') # adiciona as linhas extras criadas iris_2 = rbind(iris_2, linha_extra_1) iris_2 = rbind(iris_2, linha_extra_2) iris_2 = rbind(iris_2, linha_extra_3) # verifica resultado final View(iris_2) # exporta a base final write.csv(iris_2, file = "Iris_Alterada.csv")
E é isso por hoje! Espero que o leitor deste humilde blog tenha feito bom proveito. Creio que cobrimos muita coisa nesse tutorial. E fica como lição de casa aplicar o que foi ensinado aqui em algum base do Kaggle.
Gostou do conteúdo? Se inscreva para receber as novidades! Deixe seu e-mail em INSCREVA-SE na barra à direita, logo abaixo de pesquisar. E, por favor, não deixe de comentar, dar seu feedback e compartilhar com seus amigos. De verdade, isso faz toda a diferença. Você também pode acompanhar mais do meu trabalho seguindo a conta de Twitter @EstatSite ou por alguma das redes que você encontra em Sobre o Estatsite / Contato, como meu canal de Youtube Canal do Yukio.
Bons estudos!
Excelente este post.Muito obrigado.
Que bom que gostou Paulo! Precisando de qualquer ajuda, é só escrever!
Sensacional esse post!!! Muito obrigado por compartilhar seu conhecimento
Obrigado pelo feedback!
Show!
Obrigado, Silmar!
Não estou conseguindo cadastrar meu e-mail para receber atualizações! Não está chegando o e-mail, nem na caixa de entrada, nem no Spam. Sou iniciante no R, queria acompanhar seus posts.
Olá, Silmar. Obrigado por avisar. Realmente não sei o motivo do e-mail não estar chegando nas pessoas, mas de qualquer forma já confirmei a inscrição e todo novo post você será informado.
Forte abraço!
Excelente. Obrigado e parabéns.
Valeu Joselias!